转录组测序助力血液肿瘤精准诊断与MRD监测
随着测序技术的发展,转录组测序(RNA-seq)已成为血液肿瘤分子诊断与治疗监测的重要工具。近日,见康华美实验室报道了一例利用RNA-seq技术发现的新型CNTRL::FGFR1融合剪接变异体。
原文链接:https://doi.org/10.1007/s12308-025-00670-6
病例介绍
患者,35岁,男性,临床表现为白细胞显著增高、淋巴结肿大、肝脾肿大,骨髓及淋巴结中同时存在B淋巴母细胞、T淋巴母细胞及单核母细胞等多系细胞异常增殖。染色体核型分析发现 t(8;9)(p11;q33) 易位,经FISH验证存在FGFR1基因重排。
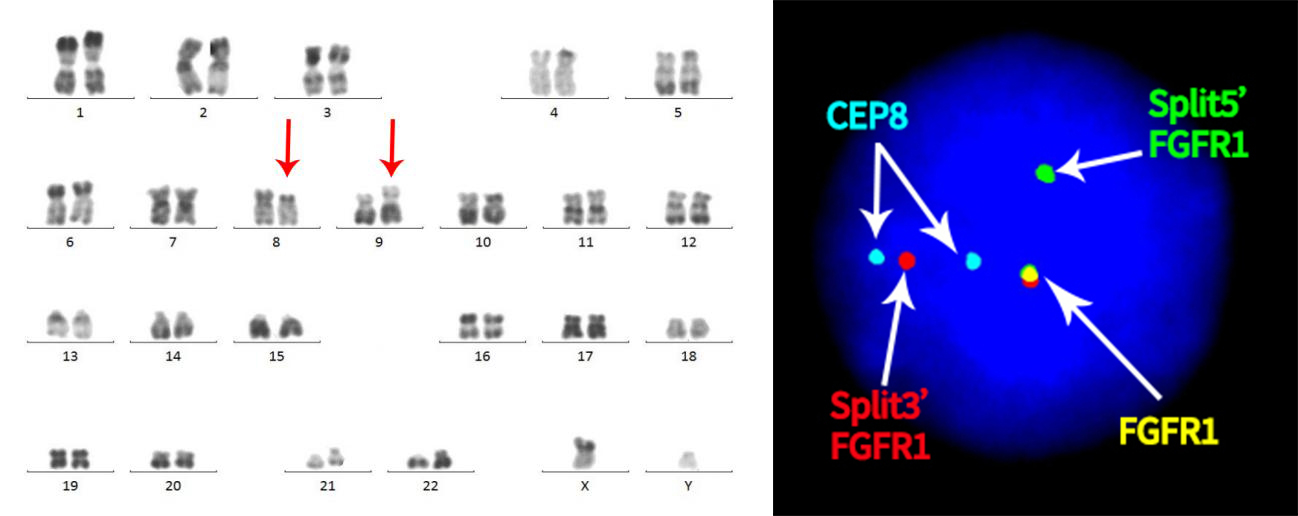
染色体核型分析(左图):46, XY, t (8;9)(p11;q33);骨髓FISH(右图):蓝色信号,8号染色体着丝粒;黄色信号,FGFR1基因;红色和绿色信号,FGFR1基因断裂产物
为明确融合伴侣基因及其具体断裂位点,实验室利用RNA-seq技术,鉴定出一种新型CNTRL::FGFR1融合剪接变异体—CNTRL基因第38号外显子与FGFR1基因第11号外显子之间形成的框内融合。该融合基因保留了FGFR1的激酶结构域,导致FGFR1酪氨酸激酶组成型激活,驱动细胞增殖失控。最终,临床诊断为伴嗜酸性粒细胞增多和FGFR1重排的髓系/淋系肿瘤(MLN-FGFR1)。
.png)
转录组测序结果显示,CNTRL基因的第38号外显子与FGFR1基因的第11号外显子发生融合
CNTRL::FGFR1是MLN-FGFR1中一种极为罕见的融合亚型,其临床特征以扁桃体肥大和持续性单核细胞增多为主,且常规化疗效果不佳,提示预后不良。在FGFR1重排阳性病例中,不同融合亚型的临床与预后特征存在异质性,因此通过RNA-seq明确具体的融合基因型,对于准确评估预后和制定治疗策略具有重要指导价值。
此外,基于融合基因的精准测序结果,可进一步建立患者个性化的监测方案,用于动态监测治疗后微小残留病灶(MRD),从而及时评估治疗反应、预警早期复发,并为调整维持或干预策略提供直接依据。
RNA-seq的核心优势
全面筛查融合基因
相比传统方法如FISH或PCR需预先设定目标,RNA-seq可一次性筛查所有可能的融合事件,大幅提升诊断效率。与DNA高通量测序相比,RNA-seq直接捕获表达后的融合转录本,避免DNA水平上因大量内含子区域或沉默融合导致的漏检,从而提高检测灵敏度和临床相关性,尤其适用于未知融合伙伴或复杂重排的发现。
精确识别断裂位点助力MRD监测
RNA-seq能精确解析融合基因的断裂位点与连接序列,基于此可设计特异性引物或探针,开发定量PCR监测方案,实现对微小残留病的高灵敏度追踪。
分析可变剪接
RNA-seq可进行可变剪接分析,能系统性识别异常剪接模式,发现新的剪接转录本。
整合突变检测与表达分析的综合价值
RNA-seq可检测基因表达水平,从而揭示血液肿瘤中过表达或沉默的关键基因。同时,RNA-seq也能识别编码区内的基因突变,尤其适用于高表达基因的变异筛查。RNA-seq的整合分析能直接关联突变与表达变化,揭示功能影响,并在单次检测中提供融合、剪接和表达等的多维信息,提升诊疗效率。
见康华美致力于提供精准的RNA-seq与MRD监测方案
标本要求
骨髓液/外周血:3-5 mL,EDTA抗凝(紫帽) / 枸酸钠抗凝(蓝帽),2-8 ℃冷藏运输;石蜡切片:厚度5 μm,组织区域≥5*5 mm,数量≥10张,常温运输。
参考文献:
Feng, X., Bai, X., Zhang, H. et al. A novel splicing variant of CNTRL::FGFR1 in myeloid/lymphoid neoplasm with eosinophilia and rearrangement of FGFR1. J Hematopathol 18, 54 (2025).